 Voir sur TensorFlow.org Voir sur TensorFlow.org |  Exécuter dans Google Colab Exécuter dans Google Colab |  Voir la source sur GitHub Voir la source sur GitHub |  Télécharger le cahier Télécharger le cahier |
Aperçu
Ce tutoriel montre comment utiliser tfio.image.decode_dicom_image dans tensorflow IO pour décoder les fichiers DICOM avec tensorflow.
Configuration et utilisation
Télécharger l'image DICOM
L'image de DICOM utilisé dans ce tutoriel est de la poitrine NIH ensemble de données de rayons X .
Le coffre NIH ensemble de données de rayons X se compose de 100.000 images dépersonnalisées de radiographies pulmonaires au format PNG, fournies par le NIH Clinical Center et peut être téléchargé via ce lien .
Google Cloud fournit également une version DICOM des images, disponibles dans Cloud Storage .
Dans ce tutoriel, vous allez télécharger un fichier échantillon de l'ensemble de données à partir du repo GitHub
- Xiaosong Wang, Yifan Peng, Le Lu, Zhiyong Lu, Mohammadhadi Bagheri, Ronald Summers, ChestX-ray8 : base de données sur les radiographies thoraciques à l'échelle de l'hôpital et points de référence sur la classification et la localisation faiblement supervisées des maladies courantes du thorax, IEEE CVPR, pp. 3462 -3471, 2017
curl -OL https://github.com/tensorflow/io/raw/master/docs/tutorials/dicom/dicom_00000001_000.dcmls -l dicom_00000001_000.dcm
% Total % Received % Xferd Average Speed Time Time Time Current
Dload Upload Total Spent Left Speed
100 164 0 164 0 0 600 0 --:--:-- --:--:-- --:--:-- 598
100 1024k 100 1024k 0 0 1915k 0 --:--:-- --:--:-- --:--:-- 1915k
-rw-rw-r-- 1 kbuilder kokoro 1049332 Nov 22 03:47 dicom_00000001_000.dcm
Installez les packages requis et redémarrez le runtime
try:
# Use the Colab's preinstalled TensorFlow 2.x
%tensorflow_version 2.x
except:
pass
pip install tensorflow-io
Décoder l'image DICOM
import matplotlib.pyplot as plt
import numpy as np
import tensorflow as tf
import tensorflow_io as tfio
image_bytes = tf.io.read_file('dicom_00000001_000.dcm')
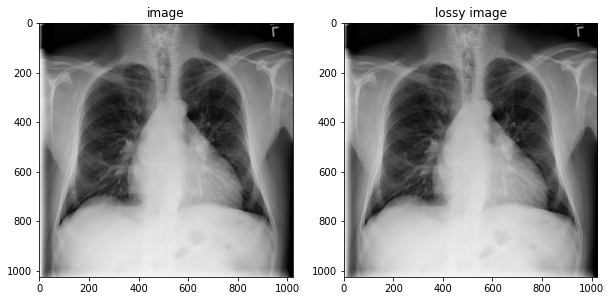
image = tfio.image.decode_dicom_image(image_bytes, dtype=tf.uint16)
skipped = tfio.image.decode_dicom_image(image_bytes, on_error='skip', dtype=tf.uint8)
lossy_image = tfio.image.decode_dicom_image(image_bytes, scale='auto', on_error='lossy', dtype=tf.uint8)
fig, axes = plt.subplots(1,2, figsize=(10,10))
axes[0].imshow(np.squeeze(image.numpy()), cmap='gray')
axes[0].set_title('image')
axes[1].imshow(np.squeeze(lossy_image.numpy()), cmap='gray')
axes[1].set_title('lossy image');
2021-11-22 03:47:53.016507: E tensorflow/stream_executor/cuda/cuda_driver.cc:271] failed call to cuInit: CUDA_ERROR_NO_DEVICE: no CUDA-capable device is detected
Décoder les métadonnées DICOM et travailler avec les balises
decode_dicom_data décode les informations d'étiquette. dicom_tags contient des informations utiles comme l'âge et le sexe du patient, de sorte que vous pouvez utiliser des balises DICOM tels que dicom_tags.PatientsAge et dicom_tags.PatientsSex . tensorflow_io emprunte la même notation de balise du package pydicom dicom.
tag_id = tfio.image.dicom_tags.PatientsAge
tag_value = tfio.image.decode_dicom_data(image_bytes,tag_id)
print(tag_value)
tf.Tensor(b'58', shape=(), dtype=string)
print(f"PatientsAge : {tag_value.numpy().decode('UTF-8')}")
PatientsAge : 58
tag_id = tfio.image.dicom_tags.PatientsSex
tag_value = tfio.image.decode_dicom_data(image_bytes,tag_id)
print(f"PatientsSex : {tag_value.numpy().decode('UTF-8')}")
PatientsSex : M
