Visualização : Explorar em Conheça seus dados
Descrição :
O CBIS-DDSM (Curated Breast Imaging Subset of DDSM) é uma versão atualizada e padronizada do Digital Database for Screening Mammography (DDSM). O DDSM é um banco de dados de 2.620 estudos de mamografia digitalizados. Ele contém casos normais, benignos e malignos com informações de patologia verificadas.
A configuração padrão é feita de patches extraídos das mamografias originais, seguindo a descrição de ( http://arxiv.org/abs/1708.09427 ), a fim de enquadrar a tarefa a resolver em uma configuração de classificação de imagem tradicional.
Página inicial : https://wiki.cancerimagingarchive.net/display/Public/CBIS-DDSM
Código-fonte :
tfds.image_classification.CuratedBreastImagingDDSMVersões :
-
2.0.1: Nova API de divisão ( https://tensorflow.org/datasets/splits ) -
3.0.0(padrão): melhor amostragem de corte ( https://github.com/tensorflow/datasets/pull/2502 )
-
Instruções de download manual : este conjunto de dados exige que você baixe os dados de origem manualmente em
download_config.manual_dir(o padrão é~/tensorflow_datasets/downloads/manual/):
Você pode baixar as imagens em https://wiki.cancerimagingarchive.net/display/Public/CBIS-DDSM
Como são necessários software e bibliotecas especiais para baixar e ler as imagens contidas no conjunto de dados, o TFDS assume que o usuário baixou os arquivos DCIM originais e os converteu em PNG.
Os seguintes comandos (ou equivalentes) devem ser usados para gerar os arquivos PNG, a fim de garantir resultados reprodutíveis:
find $DATASET_DCIM_DIR -name '*.dcm' | \
xargs -n1 -P8 -I{} bash -c 'f={}; dcmj2pnm $f | convert - ${f/.dcm/.png}'
As imagens resultantes devem ser colocadas em manual_dir , como: <manual_dir>/Mass-Training_P_01981_RIGHT_MLO_1/1.3.6.../000000.png .
Armazenado em cache automaticamente ( documentação ): Não
Chaves supervisionadas (Consulte
as_superviseddoc ):NoneCitação :
@misc{CBIS_DDSM_Citation,
doi = {10.7937/k9/tcia.2016.7o02s9cy},
url = {https://wiki.cancerimagingarchive.net/x/lZNXAQ},
author = {Sawyer-Lee, Rebecca and Gimenez, Francisco and Hoogi, Assaf and Rubin, Daniel},
title = {Curated Breast Imaging Subset of DDSM},
publisher = {The Cancer Imaging Archive},
year = {2016},
}
@article{TCIA_Citation,
author = {
K. Clark and B. Vendt and K. Smith and J. Freymann and J. Kirby and
P. Koppel and S. Moore and S. Phillips and D. Maffitt and M. Pringle and
L. Tarbox and F. Prior
},
title = { {The Cancer Imaging Archive (TCIA): Maintaining and Operating a
Public Information Repository} },
journal = {Journal of Digital Imaging},
volume = {26},
month = {December},
year = {2013},
pages = {1045-1057},
}
@article{DBLP:journals/corr/abs-1708-09427,
author = {Li Shen},
title = {End-to-end Training for Whole Image Breast Cancer Diagnosis using
An All Convolutional Design},
journal = {CoRR},
volume = {abs/1708.09427},
year = {2017},
url = {http://arxiv.org/abs/1708.09427},
archivePrefix = {arXiv},
eprint = {1708.09427},
timestamp = {Mon, 13 Aug 2018 16:48:35 +0200},
biburl = {https://dblp.org/rec/bib/journals/corr/abs-1708-09427},
bibsource = {dblp computer science bibliography, https://dblp.org}
}
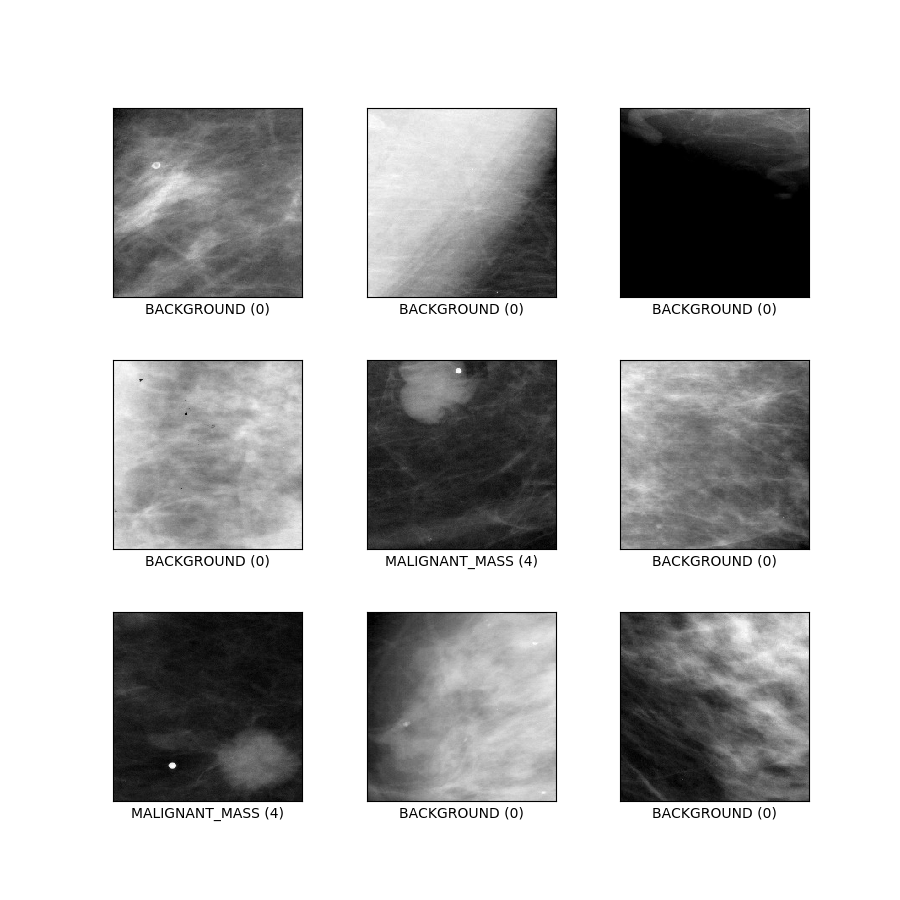
curated_breast_imaging_ddsm/patches (configuração padrão)
Descrição da configuração : Patches contendo casos de calsificação e massa, além de caminhos sem anormalidades. Projetado como uma tarefa tradicional de classificação de 5 classes.
Tamanho do download :
2.01 MiBTamanho do conjunto de dados :
801.46 MiBDivisões :
| Dividir | Exemplos |
|---|---|
'test' | 9.770 |
'train' | 49.780 |
'validation' | 5.580 |
- Estrutura de recursos :
FeaturesDict({
'id': Text(shape=(), dtype=string),
'image': Image(shape=(None, None, 1), dtype=uint8),
'label': ClassLabel(shape=(), dtype=int64, num_classes=5),
})
- Documentação do recurso :
| Recurso | Aula | Forma | Tipo D | Descrição |
|---|---|---|---|---|
| RecursosDict | ||||
| eu ia | Texto | corda | ||
| imagem | Imagem | (Nenhuma, Nenhuma, 1) | uint8 | |
| rótulo | ClassLabel | int64 |
- Figura ( tfds.show_examples ):
- Exemplos ( tfds.as_dataframe ):
curadoria_breast_imaging_ddsm/original-calc
Descrição da configuração : Imagens originais dos casos de calcificação compactadas em PNG sem perdas.
Tamanho do download :
1.06 MiBTamanho do conjunto de dados :
4.42 GiBDivisões :
| Dividir | Exemplos |
|---|---|
'test' | 284 |
'train' | 1.227 |
- Estrutura de recursos :
FeaturesDict({
'abnormalities': Sequence({
'assessment': ClassLabel(shape=(), dtype=int64, num_classes=6),
'calc_distribution': ClassLabel(shape=(), dtype=int64, num_classes=10),
'calc_type': ClassLabel(shape=(), dtype=int64, num_classes=48),
'id': int32,
'mask': Image(shape=(None, None, 1), dtype=uint8),
'pathology': ClassLabel(shape=(), dtype=int64, num_classes=3),
'subtlety': ClassLabel(shape=(), dtype=int64, num_classes=6),
}),
'breast': ClassLabel(shape=(), dtype=int64, num_classes=2),
'id': Text(shape=(), dtype=string),
'image': Image(shape=(None, None, 1), dtype=uint8),
'patient': Text(shape=(), dtype=string),
'view': ClassLabel(shape=(), dtype=int64, num_classes=2),
})
- Documentação do recurso :
| Recurso | Aula | Forma | Tipo D | Descrição |
|---|---|---|---|---|
| RecursosDict | ||||
| anormalidades | Seqüência | |||
| anormalidades/avaliação | ClassLabel | int64 | ||
| anormalidades/distribuição_calc | ClassLabel | int64 | ||
| anormalidades/calc_type | ClassLabel | int64 | ||
| anormalidades/id | tensor | int32 | ||
| anormalidades/máscara | Imagem | (Nenhuma, Nenhuma, 1) | uint8 | |
| anormalidades/patologia | ClassLabel | int64 | ||
| anormalidades/sutileza | ClassLabel | int64 | ||
| seios | ClassLabel | int64 | ||
| eu ia | Texto | corda | ||
| imagem | Imagem | (Nenhuma, Nenhuma, 1) | uint8 | |
| paciente | Texto | corda | ||
| visualizar | ClassLabel | int64 |
- Figura ( tfds.show_examples ):
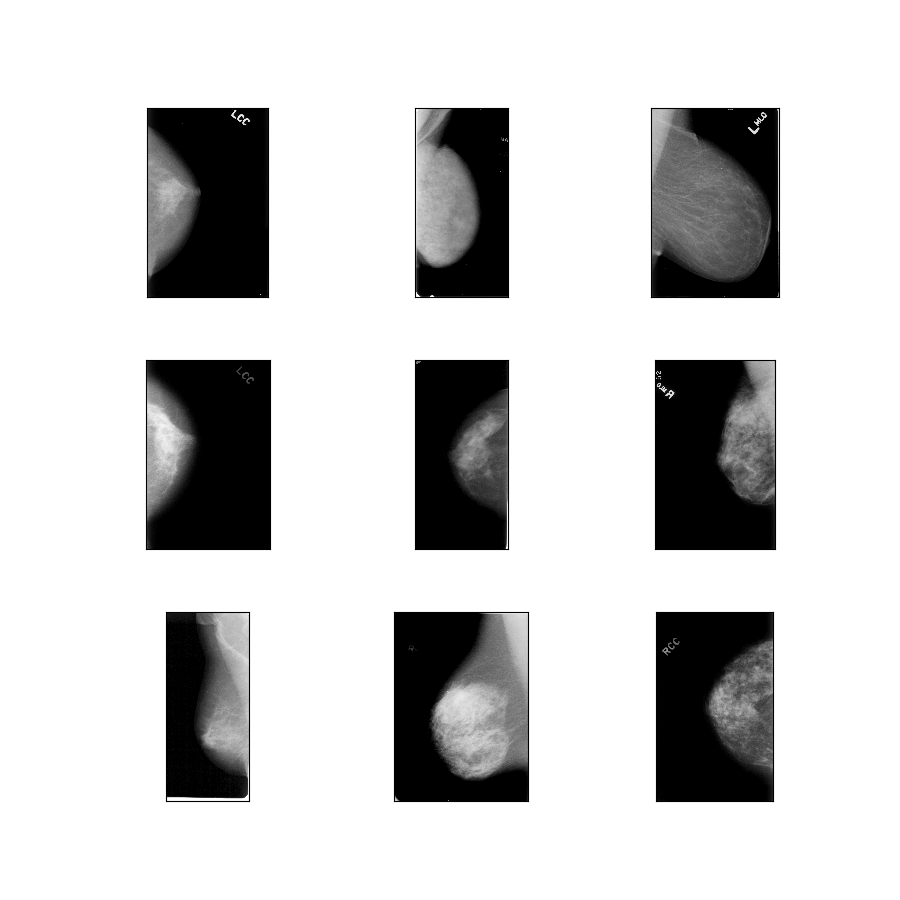
- Exemplos ( tfds.as_dataframe ):
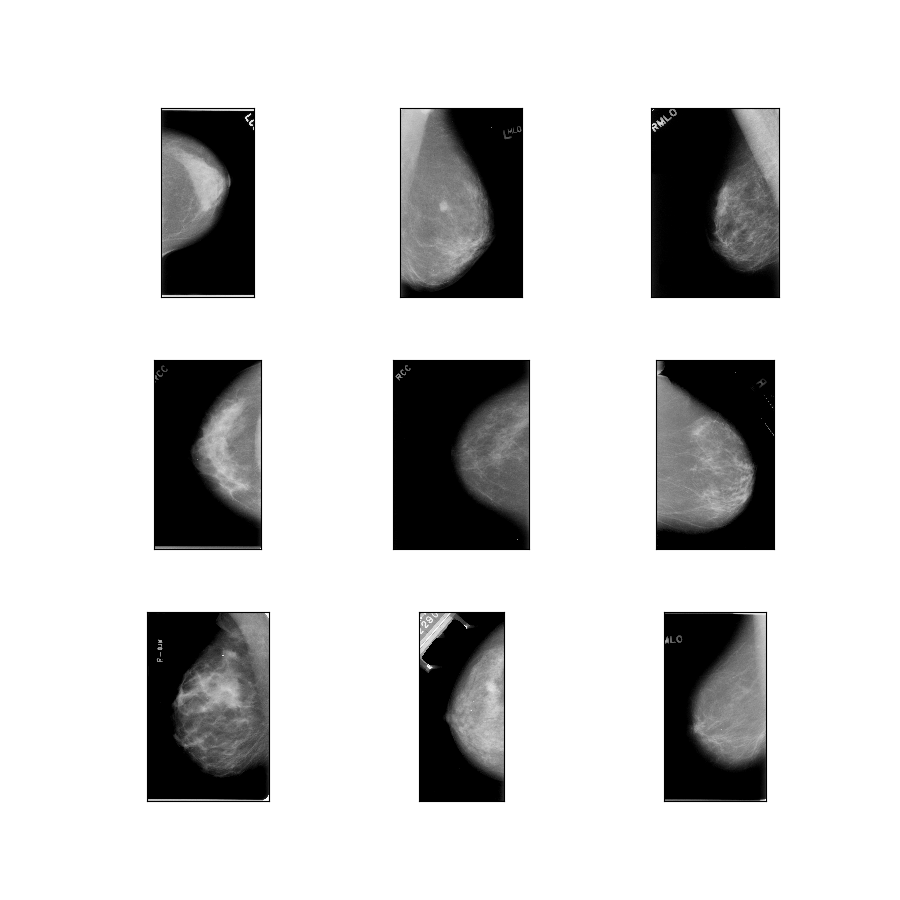
curated_breast_imaging_ddsm/original-mass
Descrição da configuração : Imagens originais dos casos em massa compactadas em PNG sem perdas.
Tamanho do download :
966.57 KiBTamanho do conjunto de dados :
4.80 GiBDivisões :
| Dividir | Exemplos |
|---|---|
'test' | 348 |
'train' | 1.166 |
- Estrutura de recursos :
FeaturesDict({
'abnormalities': Sequence({
'assessment': ClassLabel(shape=(), dtype=int64, num_classes=6),
'id': int32,
'mask': Image(shape=(None, None, 1), dtype=uint8),
'mass_margins': ClassLabel(shape=(), dtype=int64, num_classes=20),
'mass_shape': ClassLabel(shape=(), dtype=int64, num_classes=21),
'pathology': ClassLabel(shape=(), dtype=int64, num_classes=3),
'subtlety': ClassLabel(shape=(), dtype=int64, num_classes=6),
}),
'breast': ClassLabel(shape=(), dtype=int64, num_classes=2),
'id': Text(shape=(), dtype=string),
'image': Image(shape=(None, None, 1), dtype=uint8),
'patient': Text(shape=(), dtype=string),
'view': ClassLabel(shape=(), dtype=int64, num_classes=2),
})
- Documentação do recurso :
| Recurso | Aula | Forma | Tipo D | Descrição |
|---|---|---|---|---|
| RecursosDict | ||||
| anormalidades | Seqüência | |||
| anormalidades/avaliação | ClassLabel | int64 | ||
| anormalidades/id | tensor | int32 | ||
| anormalidades/máscara | Imagem | (Nenhuma, Nenhuma, 1) | uint8 | |
| anormalidades/mass_margins | ClassLabel | int64 | ||
| anormalidades/massa_forma | ClassLabel | int64 | ||
| anormalidades/patologia | ClassLabel | int64 | ||
| anormalidades/sutileza | ClassLabel | int64 | ||
| seios | ClassLabel | int64 | ||
| eu ia | Texto | corda | ||
| imagem | Imagem | (Nenhuma, Nenhuma, 1) | uint8 | |
| paciente | Texto | corda | ||
| visualizar | ClassLabel | int64 |
- Figura ( tfds.show_examples ):
- Exemplos ( tfds.as_dataframe ):

