Visualización : Explore en Know Your Data
Descripción :
El CBIS-DDSM (Subconjunto curado de imágenes mamarias de DDSM) es una versión actualizada y estandarizada de la base de datos digital para mamografías de detección (DDSM). El DDSM es una base de datos de 2620 estudios de mamografía de película escaneada. Contiene casos normales, benignos y malignos con información patológica verificada.
La configuración por defecto está hecha de parches extraídos de las mamografías originales, siguiendo la descripción de ( http://arxiv.org/abs/1708.09427 ), para enmarcar la tarea a resolver en una configuración tradicional de clasificación de imágenes.
Página de inicio : https://wiki.cancerimagingarchive.net/display/Public/CBIS-DDSM
Código fuente :
tfds.image_classification.CuratedBreastImagingDDSMVersiones :
-
2.0.1: nueva API dividida ( https://tensorflow.org/datasets/splits ) -
3.0.0(predeterminado): Mejor muestreo de recorte ( https://github.com/tensorflow/datasets/pull/2502 )
-
Instrucciones de descarga manual : este conjunto de datos requiere que descargue los datos de origen manualmente en
download_config.manual_dir(el valor predeterminado es~/tensorflow_datasets/downloads/manual/):
Puede descargar las imágenes desde https://wiki.cancerimagingarchive.net/display/Public/CBIS-DDSM
Debido a que se necesitan software y bibliotecas especiales para descargar y leer las imágenes contenidas en el conjunto de datos, TFDS asume que el usuario descargó los archivos DCIM originales y los convirtió a PNG.
Se deben utilizar los siguientes comandos (o equivalentes) para generar los archivos PNG, con el fin de garantizar resultados reproducibles:
find $DATASET_DCIM_DIR -name '*.dcm' | \
xargs -n1 -P8 -I{} bash -c 'f={}; dcmj2pnm $f | convert - ${f/.dcm/.png}'
Las imágenes resultantes deben colocarse en manual_dir , como: <manual_dir>/Mass-Training_P_01981_RIGHT_MLO_1/1.3.6.../000000.png .
Almacenamiento automático en caché ( documentación ): No
Claves supervisadas (Ver
as_superviseddoc ):NoneCita :
@misc{CBIS_DDSM_Citation,
doi = {10.7937/k9/tcia.2016.7o02s9cy},
url = {https://wiki.cancerimagingarchive.net/x/lZNXAQ},
author = {Sawyer-Lee, Rebecca and Gimenez, Francisco and Hoogi, Assaf and Rubin, Daniel},
title = {Curated Breast Imaging Subset of DDSM},
publisher = {The Cancer Imaging Archive},
year = {2016},
}
@article{TCIA_Citation,
author = {
K. Clark and B. Vendt and K. Smith and J. Freymann and J. Kirby and
P. Koppel and S. Moore and S. Phillips and D. Maffitt and M. Pringle and
L. Tarbox and F. Prior
},
title = { {The Cancer Imaging Archive (TCIA): Maintaining and Operating a
Public Information Repository} },
journal = {Journal of Digital Imaging},
volume = {26},
month = {December},
year = {2013},
pages = {1045-1057},
}
@article{DBLP:journals/corr/abs-1708-09427,
author = {Li Shen},
title = {End-to-end Training for Whole Image Breast Cancer Diagnosis using
An All Convolutional Design},
journal = {CoRR},
volume = {abs/1708.09427},
year = {2017},
url = {http://arxiv.org/abs/1708.09427},
archivePrefix = {arXiv},
eprint = {1708.09427},
timestamp = {Mon, 13 Aug 2018 16:48:35 +0200},
biburl = {https://dblp.org/rec/bib/journals/corr/abs-1708-09427},
bibsource = {dblp computer science bibliography, https://dblp.org}
}
curated_breast_imaging_ddsm/patches (configuración predeterminada)
Descripción de la configuración : parches que contienen casos de calsificación y masa, además de rutas sin anomalías. Diseñado como una tarea de clasificación tradicional de 5 clases.
Tamaño de la descarga :
2.01 MiBTamaño del conjunto de datos :
801.46 MiBDivisiones :
| Dividir | Ejemplos |
|---|---|
'test' | 9,770 |
'train' | 49,780 |
'validation' | 5,580 |
- Estructura de características :
FeaturesDict({
'id': Text(shape=(), dtype=string),
'image': Image(shape=(None, None, 1), dtype=uint8),
'label': ClassLabel(shape=(), dtype=int64, num_classes=5),
})
- Documentación de características :
| Característica | Clase | Forma | Tipo D | Descripción |
|---|---|---|---|---|
| CaracterísticasDict | ||||
| identificación | Texto | cadena | ||
| imagen | Imagen | (Ninguno, Ninguno, 1) | uint8 | |
| etiqueta | Etiqueta de clase | int64 |
- Figura ( tfds.show_examples ):
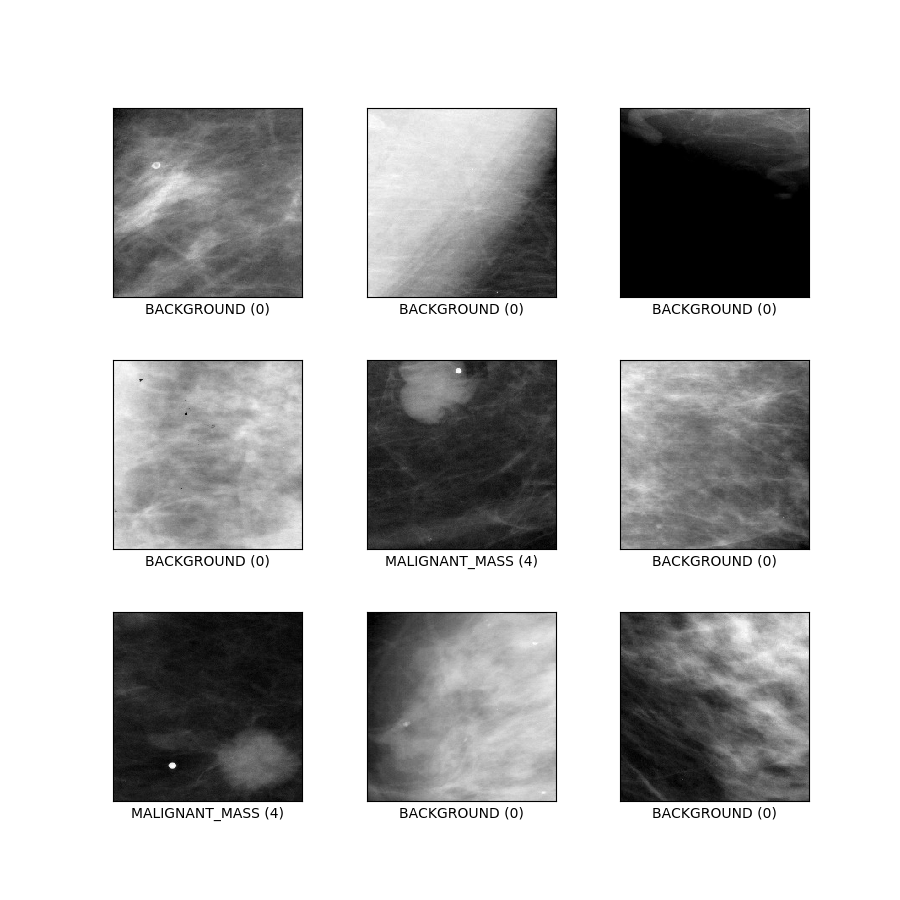
- Ejemplos ( tfds.as_dataframe ):
curated_breast_imaging_ddsm/original-calc
Descripción de la configuración : imágenes originales de los casos de calcificación comprimidas en PNG sin pérdidas.
Tamaño de la descarga :
1.06 MiBTamaño del conjunto de datos :
4.42 GiBDivisiones :
| Dividir | Ejemplos |
|---|---|
'test' | 284 |
'train' | 1,227 |
- Estructura de características :
FeaturesDict({
'abnormalities': Sequence({
'assessment': ClassLabel(shape=(), dtype=int64, num_classes=6),
'calc_distribution': ClassLabel(shape=(), dtype=int64, num_classes=10),
'calc_type': ClassLabel(shape=(), dtype=int64, num_classes=48),
'id': int32,
'mask': Image(shape=(None, None, 1), dtype=uint8),
'pathology': ClassLabel(shape=(), dtype=int64, num_classes=3),
'subtlety': ClassLabel(shape=(), dtype=int64, num_classes=6),
}),
'breast': ClassLabel(shape=(), dtype=int64, num_classes=2),
'id': Text(shape=(), dtype=string),
'image': Image(shape=(None, None, 1), dtype=uint8),
'patient': Text(shape=(), dtype=string),
'view': ClassLabel(shape=(), dtype=int64, num_classes=2),
})
- Documentación de características :
| Característica | Clase | Forma | Tipo D | Descripción |
|---|---|---|---|---|
| CaracterísticasDict | ||||
| anormalidades | Secuencia | |||
| anomalías/evaluación | Etiqueta de clase | int64 | ||
| anormalidades/calc_distribution | Etiqueta de clase | int64 | ||
| anomalías/calc_type | Etiqueta de clase | int64 | ||
| anomalías/identificación | Tensor | int32 | ||
| anomalías/máscara | Imagen | (Ninguno, Ninguno, 1) | uint8 | |
| anormalidades/patología | Etiqueta de clase | int64 | ||
| anormalidades/sutilezas | Etiqueta de clase | int64 | ||
| mama | Etiqueta de clase | int64 | ||
| identificación | Texto | cadena | ||
| imagen | Imagen | (Ninguno, Ninguno, 1) | uint8 | |
| paciente | Texto | cadena | ||
| vista | Etiqueta de clase | int64 |
- Figura ( tfds.show_examples ):
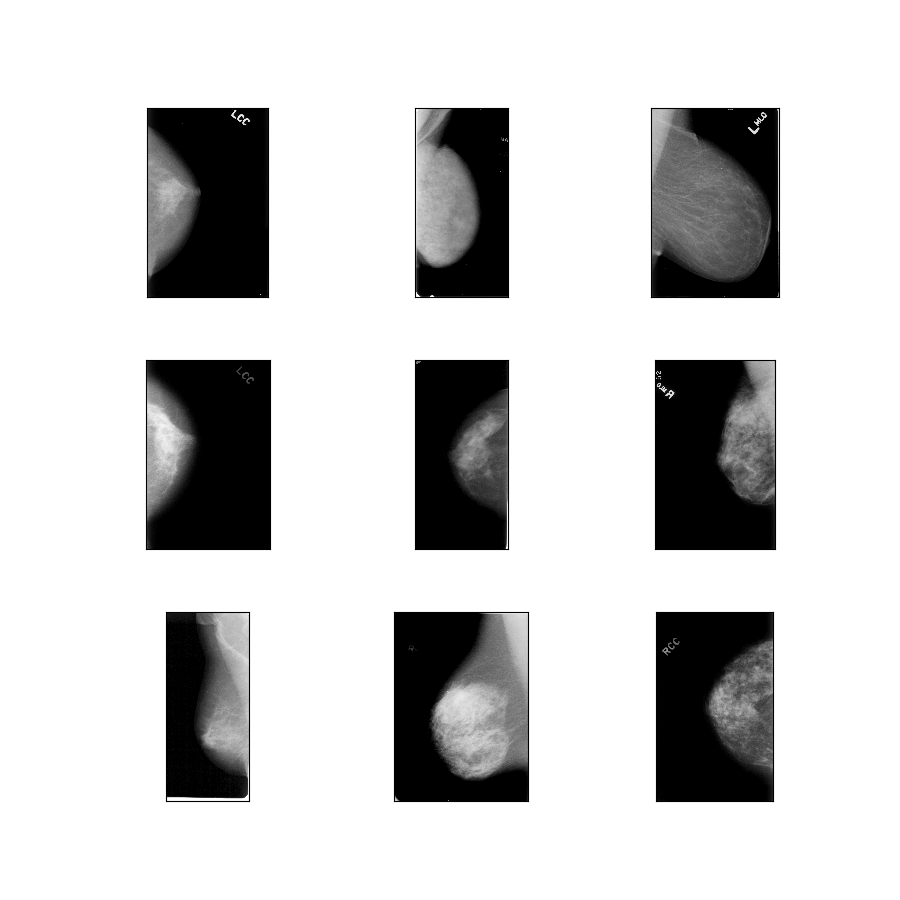
- Ejemplos ( tfds.as_dataframe ):
curated_breast_imaging_ddsm/original-mass
Descripción de la configuración : imágenes originales de los casos masivos comprimidas en PNG sin pérdidas.
Tamaño de la descarga :
966.57 KiBTamaño del conjunto de datos :
4.80 GiBDivisiones :
| Dividir | Ejemplos |
|---|---|
'test' | 348 |
'train' | 1,166 |
- Estructura de características :
FeaturesDict({
'abnormalities': Sequence({
'assessment': ClassLabel(shape=(), dtype=int64, num_classes=6),
'id': int32,
'mask': Image(shape=(None, None, 1), dtype=uint8),
'mass_margins': ClassLabel(shape=(), dtype=int64, num_classes=20),
'mass_shape': ClassLabel(shape=(), dtype=int64, num_classes=21),
'pathology': ClassLabel(shape=(), dtype=int64, num_classes=3),
'subtlety': ClassLabel(shape=(), dtype=int64, num_classes=6),
}),
'breast': ClassLabel(shape=(), dtype=int64, num_classes=2),
'id': Text(shape=(), dtype=string),
'image': Image(shape=(None, None, 1), dtype=uint8),
'patient': Text(shape=(), dtype=string),
'view': ClassLabel(shape=(), dtype=int64, num_classes=2),
})
- Documentación de características :
| Característica | Clase | Forma | Tipo D | Descripción |
|---|---|---|---|---|
| CaracterísticasDict | ||||
| anormalidades | Secuencia | |||
| anomalías/evaluación | Etiqueta de clase | int64 | ||
| anomalías/identificación | Tensor | int32 | ||
| anomalías/máscara | Imagen | (Ninguno, Ninguno, 1) | uint8 | |
| anormalidades/mass_margins | Etiqueta de clase | int64 | ||
| anomalías/masa_forma | Etiqueta de clase | int64 | ||
| anormalidades/patología | Etiqueta de clase | int64 | ||
| anormalidades/sutilezas | Etiqueta de clase | int64 | ||
| mama | Etiqueta de clase | int64 | ||
| identificación | Texto | cadena | ||
| imagen | Imagen | (Ninguno, Ninguno, 1) | uint8 | |
| paciente | Texto | cadena | ||
| vista | Etiqueta de clase | int64 |
- Figura ( tfds.show_examples ):
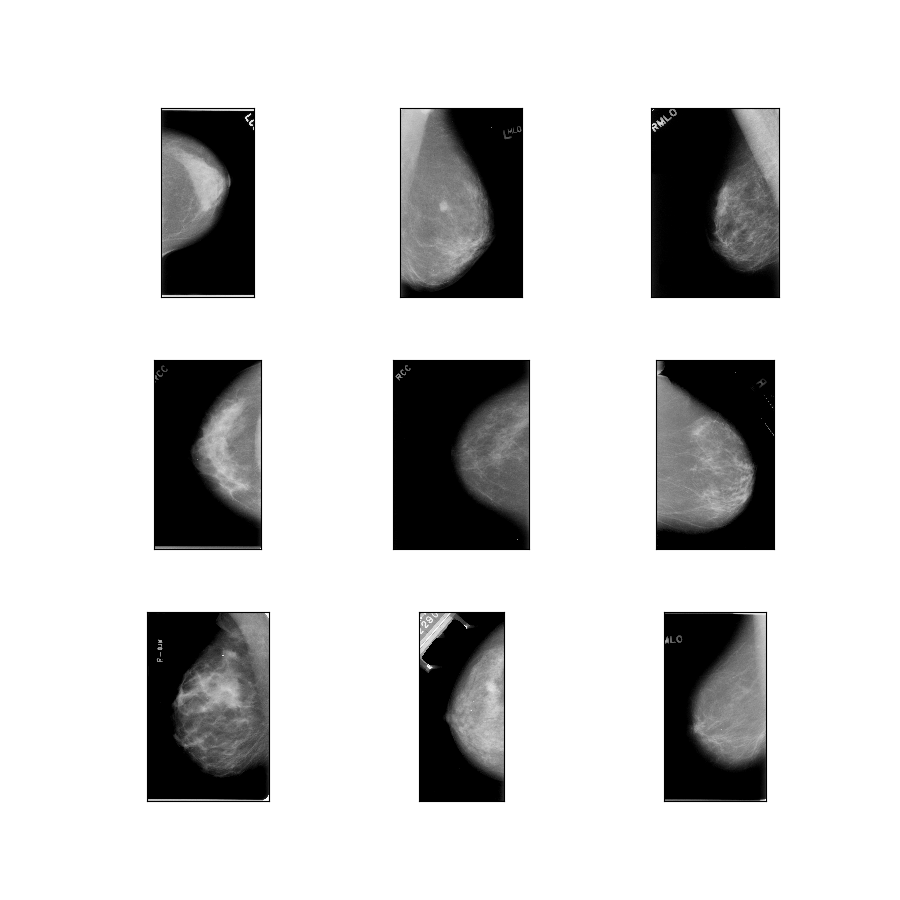
- Ejemplos ( tfds.as_dataframe ):

