- Opis :
Zbiór danych dotyczących malarii zawiera w sumie 27 558 obrazów komórek, w tym równe przypadki komórek zakażonych i niezainfekowanych, z obrazów cienkich preparatów rozmazu krwi podzielonych na segmenty komórek.
Dodatkowa dokumentacja : Eksploruj w dokumentach z kodem
Strona główna : https://lhncbc.nlm.nih.gov/publication/pub9932
Kod źródłowy :
tfds.datasets.malaria.BuilderWersje :
-
1.0.0(domyślnie): Brak informacji o wersji.
-
Rozmiar pobierania :
337.08 MiBRozmiar zbioru danych :
317.62 MiBAutomatyczne buforowanie ( dokumentacja ): Nie
Podziały :
| Podział | Przykłady |
|---|---|
'train' | 27558 |
- Struktura funkcji :
FeaturesDict({
'image': Image(shape=(None, None, 3), dtype=uint8),
'label': ClassLabel(shape=(), dtype=int64, num_classes=2),
})
- Dokumentacja funkcji :
| Funkcja | Klasa | Kształt | Typ D | Opis |
|---|---|---|---|---|
| FunkcjeDykt | ||||
| obraz | Obraz | (Brak, Brak, 3) | uint8 | |
| etykieta | Etykieta klasy | int64 |
Klucze nadzorowane (zobacz dokument
as_supervised):('image', 'label')Rysunek ( tfds.show_examples ):
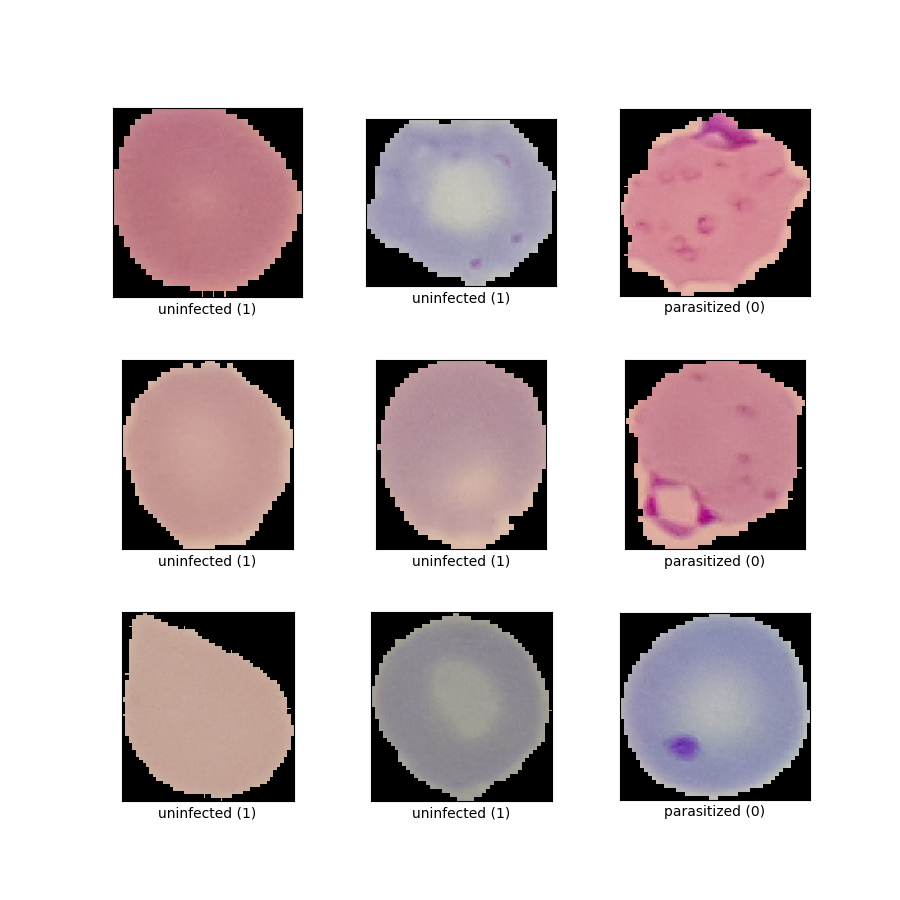
- Przykłady ( tfds.as_dataframe ):
- Cytat :
@article{rajaraman2018pre,
title={Pre-trained convolutional neural networks as feature extractors toward
improved malaria parasite detection in thin blood smear images},
author={Rajaraman, Sivaramakrishnan and Antani, Sameer K and Poostchi, Mahdieh
and Silamut, Kamolrat and Hossain, Md A and Maude, Richard J and Jaeger,
Stefan and Thoma, George R},
journal={PeerJ},
volume={6},
pages={e4568},
year={2018},
publisher={PeerJ Inc.}
}

