- विवरण :
'ओजीबीजी-एमओएलपीसीबीए' पबकेम बायोएसे से नमूना लिया गया एक आणविक डेटासेट है। यह ओपन ग्राफ़ बेंचमार्क (ओजीबी) से एक ग्राफ़ भविष्यवाणी डेटासेट है।
यह डेटासेट प्रायोगिक है, और एपीआई भविष्य के रिलीज में परिवर्तन के अधीन है।
डेटासेट के नीचे दिए गए विवरण को ओजीबी पेपर से अनुकूलित किया गया है:
इनपुट प्रारूप
RDKit ([1]) का उपयोग करके सभी अणुओं को पूर्व-संसाधित किया जाता है।
- प्रत्येक ग्राफ एक अणु का प्रतिनिधित्व करता है, जहां नोड परमाणु होते हैं, और किनारे रासायनिक बंधन होते हैं।
- इनपुट नोड विशेषताएं 9-आयामी हैं, जिनमें परमाणु संख्या और चिरायता शामिल है, साथ ही साथ अन्य अतिरिक्त परमाणु विशेषताएं जैसे कि औपचारिक प्रभार और परमाणु रिंग में है या नहीं।
- इनपुट एज विशेषताएं 3-आयामी हैं, जिसमें बॉन्ड प्रकार, बॉन्ड स्टीरियोकेमिस्ट्री, साथ ही एक अतिरिक्त बॉन्ड फीचर है जो दर्शाता है कि बॉन्ड संयुग्मित है या नहीं।
सभी सुविधाओं का सटीक विवरण https://github.com/snap-stanford/ogb/blob/master/ogb/utils/features.py पर उपलब्ध है।
भविष्यवाणी
कार्य 128 विभिन्न जैविक गतिविधियों (निष्क्रिय/सक्रिय) की भविष्यवाणी करना है। इन लक्ष्यों के बारे में अधिक विवरण के लिए [2] और [3] देखें। प्रत्येक अणु पर सभी लक्ष्य लागू नहीं होते हैं: लापता लक्ष्य NaNs द्वारा इंगित किए जाते हैं।
संदर्भ
[1]: ग्रेग लैन्ड्रम, एट अल। 'आरडीकिट: ओपन-सोर्स केमिनफॉरमैटिक्स'। यूआरएल: https://github.com/rdkit/rdkit
[2]: भरत रामसुंदर, स्टीवन केर्न्स, पैट्रिक रिले, डेल वेबस्टर, डेविड कोनरडिंग और विजय पांडे। 'मैसिवली मल्टीटास्क नेटवर्क्स फॉर ड्रग डिस्कवरी'। यूआरएल: https://arxiv.org/pdf/1502.02072.pdf
[3]: झेनकिन वू, भरत रामसुंदर, इवान एन फ़िनबर्ग, जोसेफ गोम्स, कालेब जिनीसे, अनीश एस. पप्पू, कार्ल लेस्विंग और विजय पांडे। मॉलिक्यूलनेट: मॉलिक्यूलर मशीन लर्निंग के लिए एक बेंचमार्क। केमिकल साइंस, 9(2):513-530, 2018।
स्रोत कोड :
tfds.datasets.ogbg_molpcba.Builderसंस्करण :
-
0.1.0: प्रयोगात्मक एपीआई की प्रारंभिक रिलीज। -
0.1.1: प्रत्येक ग्राफ में किनारों की संख्या को स्पष्ट रूप से प्रदर्शित करता है। -
0.1.2: ग्राफविजुअलाइज़र के लिए मेटाडेटा फ़ील्ड जोड़ें। -
0.1.3(डिफ़ॉल्ट): व्यक्तिगत कार्यों के नाम के लिए मेटाडेटा फ़ील्ड जोड़ें।
-
डाउनलोड आकार :
37.70 MiBडेटासेट का आकार :
822.53 MiBऑटो-कैश्ड ( दस्तावेज़ीकरण ): नहीं
विभाजन :
| विभाजित करना | उदाहरण |
|---|---|
'test' | 43,793 |
'train' | 350,343 |
'validation' | 43,793 |
- फ़ीचर संरचना :
FeaturesDict({
'edge_feat': Tensor(shape=(None, 3), dtype=float32),
'edge_index': Tensor(shape=(None, 2), dtype=int64),
'labels': Tensor(shape=(128,), dtype=float32),
'node_feat': Tensor(shape=(None, 9), dtype=float32),
'num_edges': Tensor(shape=(None,), dtype=int64),
'num_nodes': Tensor(shape=(None,), dtype=int64),
})
- फ़ीचर दस्तावेज़ीकरण :
| विशेषता | कक्षा | आकार | डीटाइप | विवरण |
|---|---|---|---|---|
| विशेषताएं डिक्ट | ||||
| edge_feat | टेन्सर | (कोई नहीं, 3) | फ्लोट32 | |
| edge_index | टेन्सर | (कोई नहीं, 2) | int64 | |
| लेबल | टेन्सर | (128,) | फ्लोट32 | |
| node_feat | टेन्सर | (कोई नहीं, 9) | फ्लोट32 | |
| num_edges | टेन्सर | (कोई नहीं,) | int64 | |
| num_nodes | टेन्सर | (कोई नहीं,) | int64 |
पर्यवेक्षित कुंजियाँ (
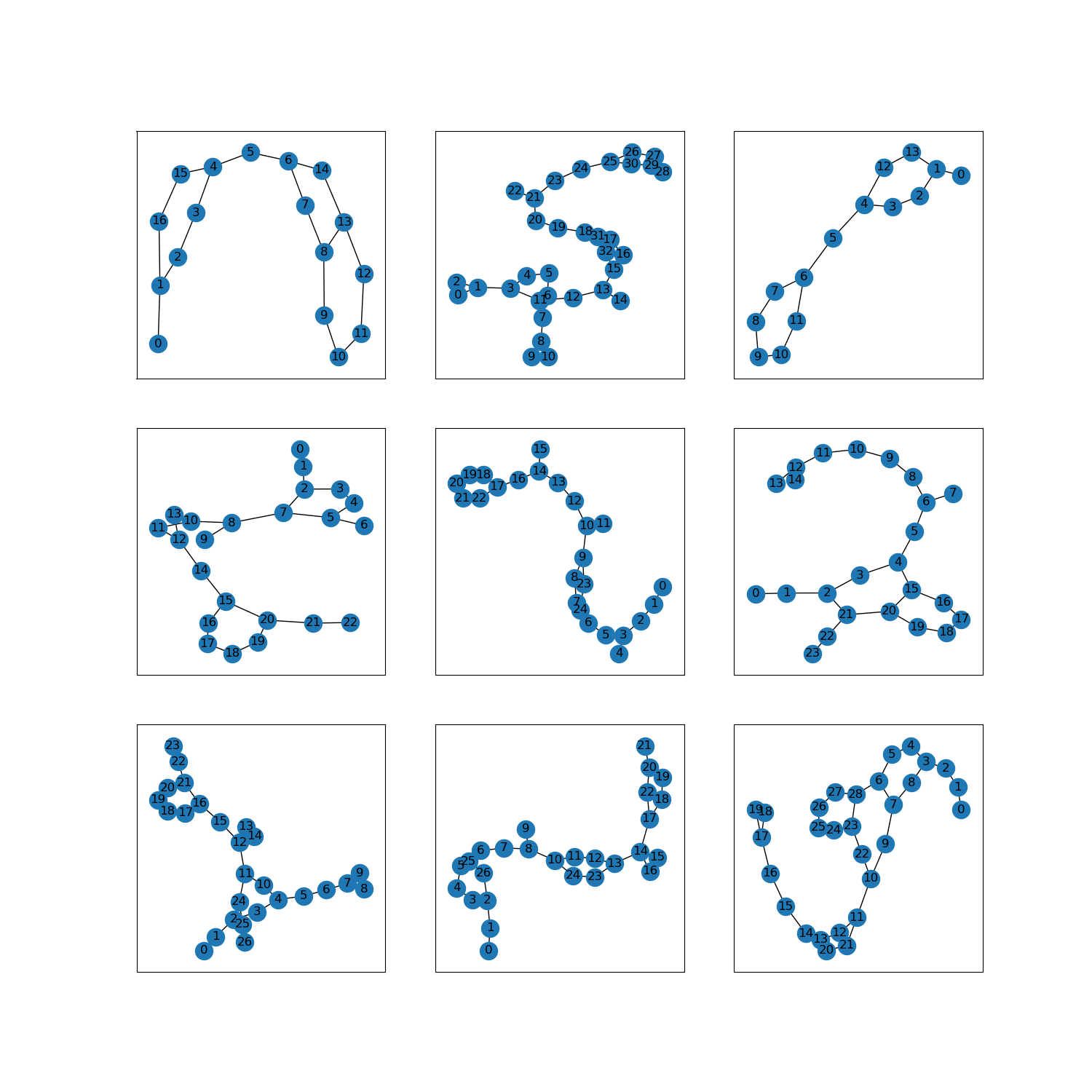
as_superviseddoc देखें):Noneचित्र ( tfds.show_examples ):
- उदाहरण ( tfds.as_dataframe ):
- उद्धरण :
@inproceedings{DBLP:conf/nips/HuFZDRLCL20,
author = {Weihua Hu and
Matthias Fey and
Marinka Zitnik and
Yuxiao Dong and
Hongyu Ren and
Bowen Liu and
Michele Catasta and
Jure Leskovec},
editor = {Hugo Larochelle and
Marc Aurelio Ranzato and
Raia Hadsell and
Maria{-}Florina Balcan and
Hsuan{-}Tien Lin},
title = {Open Graph Benchmark: Datasets for Machine Learning on Graphs},
booktitle = {Advances in Neural Information Processing Systems 33: Annual Conference
on Neural Information Processing Systems 2020, NeurIPS 2020, December
6-12, 2020, virtual},
year = {2020},
url = {https://proceedings.neurips.cc/paper/2020/hash/fb60d411a5c5b72b2e7d3527cfc84fd0-Abstract.html},
timestamp = {Tue, 19 Jan 2021 15:57:06 +0100},
biburl = {https://dblp.org/rec/conf/nips/HuFZDRLCL20.bib},
bibsource = {dblp computer science bibliography, https://dblp.org}
}

